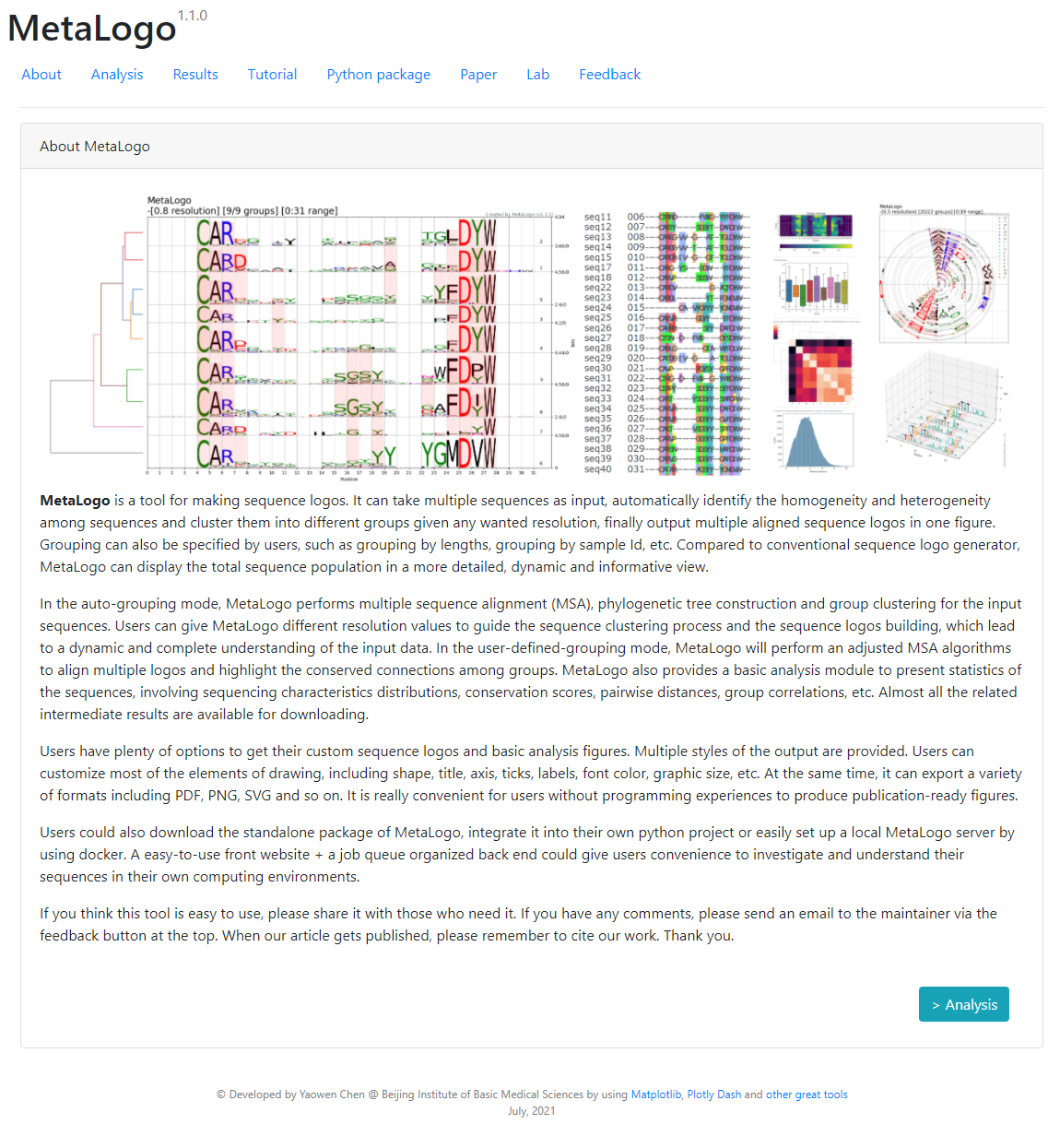
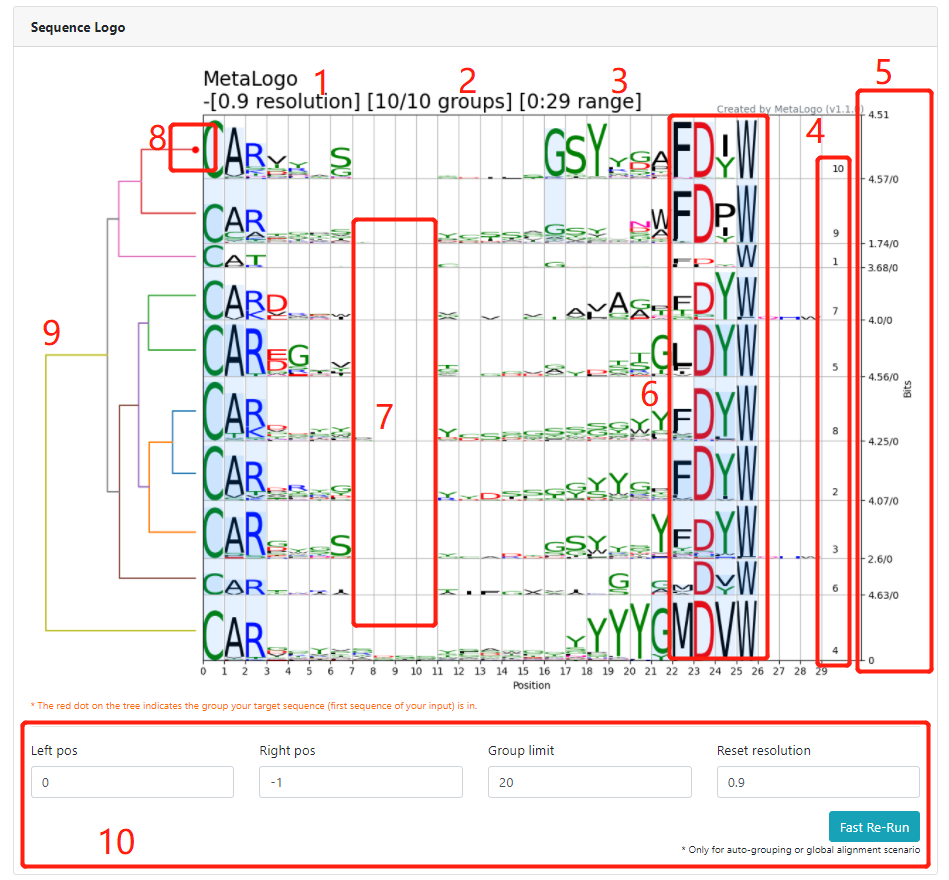
Recently, I have developed a tool named MetaLogo, aimed to make sequence logos for multiple sets of sequences.
继续阅读Make sequence logo for heterogeneous sequences or sequences with unequal length using MetaLogo?
Recently, I have developed a tool named MetaLogo, aimed to make sequence logos for multiple sets of sequences.


继续阅读Make sequence logo for heterogeneous sequences or sequences with unequal length using MetaLogo?
在生物医学研究中,生存分析 是非常重要和常见的分析方法。本文对 生存分析 中的Kaplan–Meier 模型、Cox 比例风险模型进行了简要而详尽的概述,帮助大家更好的理解生存分析等相关概念。本文适用于生物医学专业初学者以及对生存分析感兴趣的非专业人士。
首先,简单描述一下 生存分析 的使用场景,已经熟悉的同学可以选择直接跳过。 生存分析 经常用在癌症等疾病的研究中,例如在对某种抗癌药物做临床试验时,会首先筛选一部分癌症患者随机分为两组,一组服用该试验药物,一组服用对照药物,服药后开始统计每个患者从服药一直到死亡的生存时间,通过考察两组之间的病人在生存时间上是否有统计学差异来判断试验药物是否有效。在这里,死亡是整个实验中重点观测的事件,即 event。对于每个病人,需要记录他们发生该事件的具体时间。因此, 生存分析 可以抽象概述为,研究在不同条件下,特定事件发生与时间的关系是否存在差异。这些具体事件可以是死亡,也可以是肿瘤转移、复发、病人出院、重新入院等任何可以明确识别的事件,而不同条件即为不同的分组依据,可以是年龄、性别、地域、某个基因表达量的高低、某个突变的携带与否等等。下图是钟南山院士在对欧洲呼吸学会针对 Covid-19 的报告中提到的研究结果,他们对湖北省内和省外的病人从开始症状到入院时间做了分析,从发生症状开始,入院则是我们刚才讲的 event 事件,而湖北省内外则是不同的分组条件。图中还提到,他们使用 Cox 模型对地理进行了校正,这也是我们在这篇文章中后续要讲到的内容。对钟南山报告感兴趣的同学可以访问此链接 进行查看。
PyTorch 是深度学习领域著名的开发框架,本文将介绍一个完整的代码样例,从使用自定义数据开始,直到评估训练模型结束,旨在为和笔者一样的入门者提供一份可参考的样例。本文使用的神经网络模型主要为 CNN,输入数据为蛋白质序列,每一条蛋白序列通过实验可测得其某指标(Y)的数值,我们希望通过已知的蛋白序列和其对应的 Y 值,预测新序列的Y值。阅读该样例需要对 python 包 pandas 和 numpy 有一定的熟悉。
首先,简单看一下我们的数据情况。

其中 aa 一列即代表蛋白质序列,y 即代表我们需要训练的目标值。