PyTorch 是深度学习领域著名的开发框架,本文将介绍一个完整的代码样例,从使用自定义数据开始,直到评估训练模型结束,旨在为和笔者一样的入门者提供一份可参考的样例。本文使用的神经网络模型主要为 CNN,输入数据为蛋白质序列,每一条蛋白序列通过实验可测得其某指标(Y)的数值,我们希望通过已知的蛋白序列和其对应的 Y 值,预测新序列的Y值。阅读该样例需要对 python 包 pandas 和 numpy 有一定的熟悉。
首先,简单看一下我们的数据情况。

其中 aa 一列即代表蛋白质序列,y 即代表我们需要训练的目标值。
首先,我们需要对输入序列进行编码。有两种思路,一种是常用的 one-hot encoding,即每个氨基酸残基用一个20维的向量表示,向量中的20个元素只有 1 个位置为 1,其他元素均为 0。因为自然界只有 20 种氨基酸,所以用 20 个这样的向量( 1所在位置不同)即可表示所有的氨基酸残基。 举个例子,残基 A 可以用 [1,0,0,0,…,0]代表,向量种的 … 代表多个 0 的重复;残基 R 可以用 [0,1,0,0,…,0]代表;那么氨基酸序列 ARRAR 则可以编码为以下矩阵: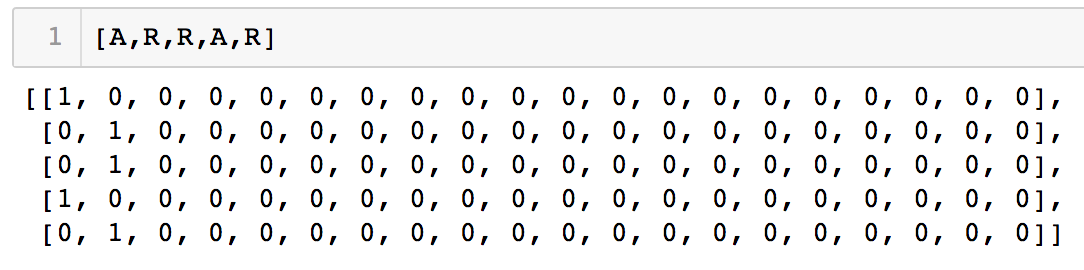
但 one-hot encoding 编码对于蛋白质序列来说,存在缺点,即没有携带残基之间相似性但信息。也就是说不同但残基之间存在着一定但关系,有的残基之间更接近,而有的残基则相差较多。我们在这里可以引入 BLOSUM 信息,它包含每两个残基之间的比较分数。一种 BLOSUM 矩阵如下图所示:
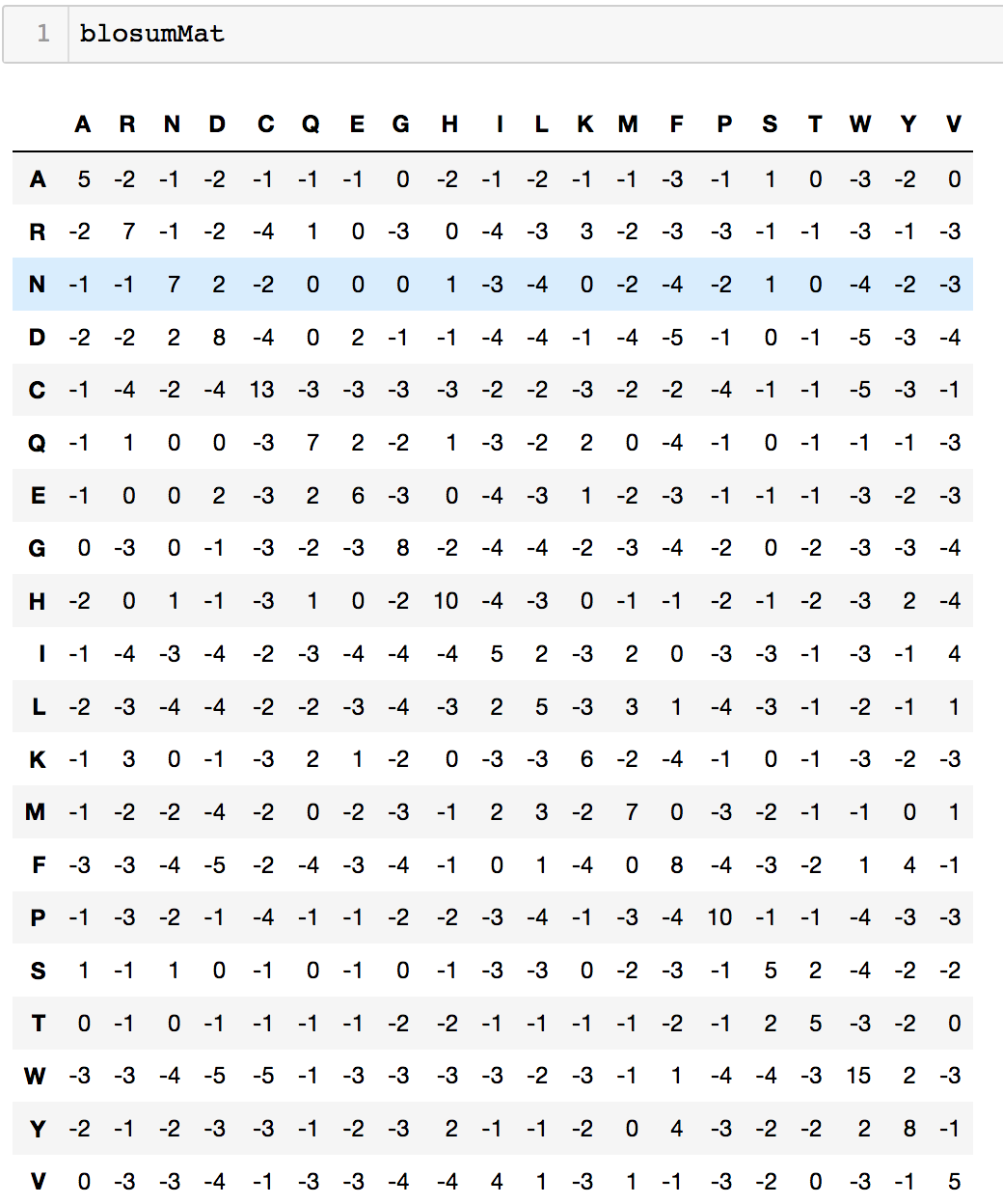
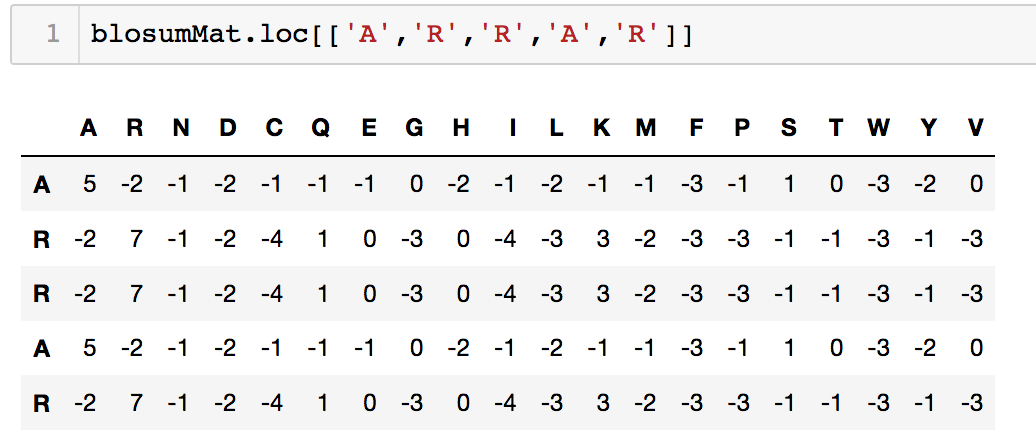
这样,我们便可以用每个残基和其他所有残基的BLOSUM分数组成的向量来编码该残基,简单来说,在上图中残基可以用其对应的一行数值来编码。之前的ARRAR序列则可以编码为:
在明确 input 编码之后,我们遇到另一个问题。我们知道,使用 CNN 网络时,需要输入长度固定,对于不同长度的氨基酸序列如何处理?有一些比较 tricky 的方法,但本文目标是为了帮助初学者跑通 PyTorch 但第一个样例,所以这里我们筛选固定长度的序列来简化模型。我们通过如下代码来得到 input 矩阵和目标值向量:
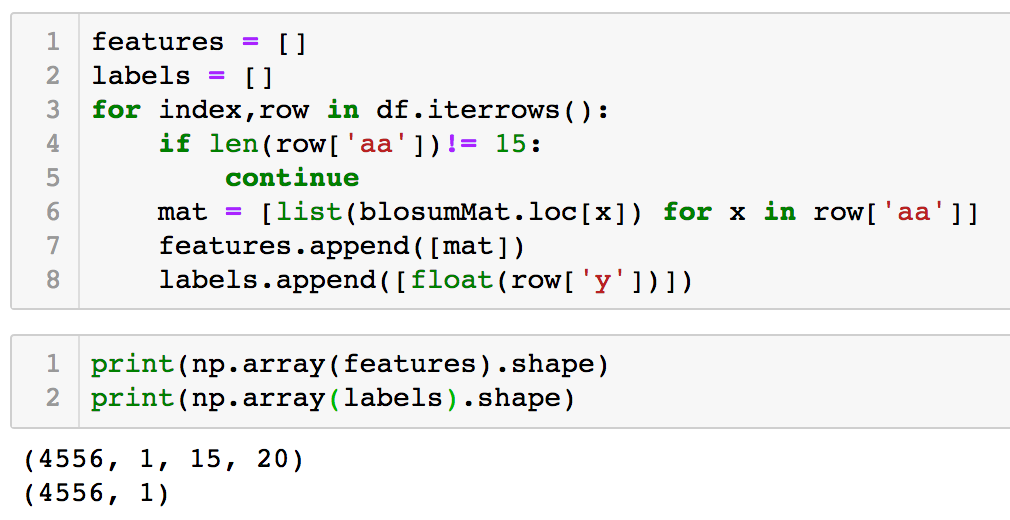
从代码中可以看到,我们选取了所有长度为 15 的氨基酸序列。
features 为输入矩阵,其 shape 为 (4556,1,15,20),其中 4556 为序列个数,1 代表的是 channel 个数,如图片数据集中的 RGB channe,这里是一维序列,所以 channel 为 1。为保持一致性,这里的 channel 维度不可忽略;15 * 20 即为编码后的序列矩阵。
labels 即所有的 Y 值向量,shape 为 4556 * 1。
构建好原始数据矩阵后,我们将其转变为 PyTorch 的 dataset 数据结构以便后续使用。
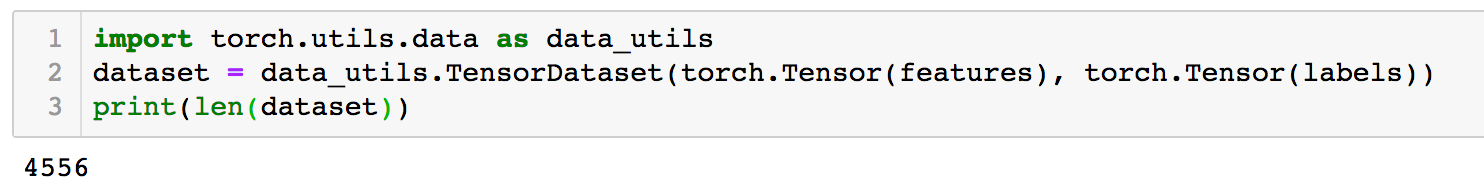
接下来,我们需要将数据分为将数据分为训练集和测试集。本文中使用的训练集和测试集比例为 7: 3,即 70% 的数据用来训练,30% 的数据用来作为测试集合。这里不设 dev 集,是为了简化流程。
我们将使用到 PyTorch 的 SubsetRandomSampler 和 DataLoader。首先,了解以下 SubsetRandomSampler 的作用:
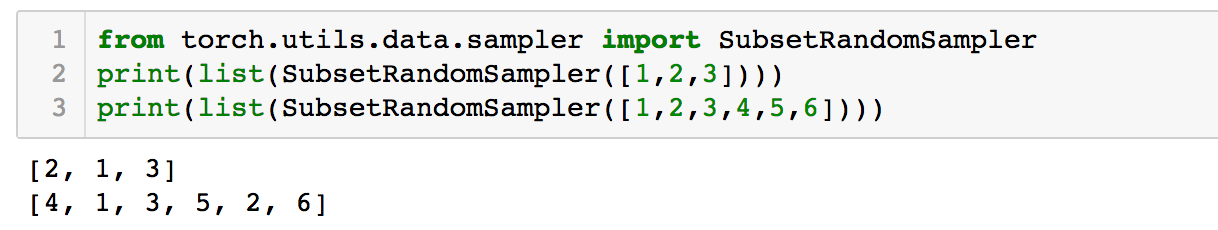
可见,subsetRandomSampler 默认是对一个数组的随机重排。具体可查看官方文档。 使用RandomSampler 而非自定义随机编号的一个理由是,在后续的每个 epoch训练中,RandomSampler 可以每次随机打乱,而非固定顺序。
因此,我们可以构建好训练集和测试集的编号,进而生成 Sampler。
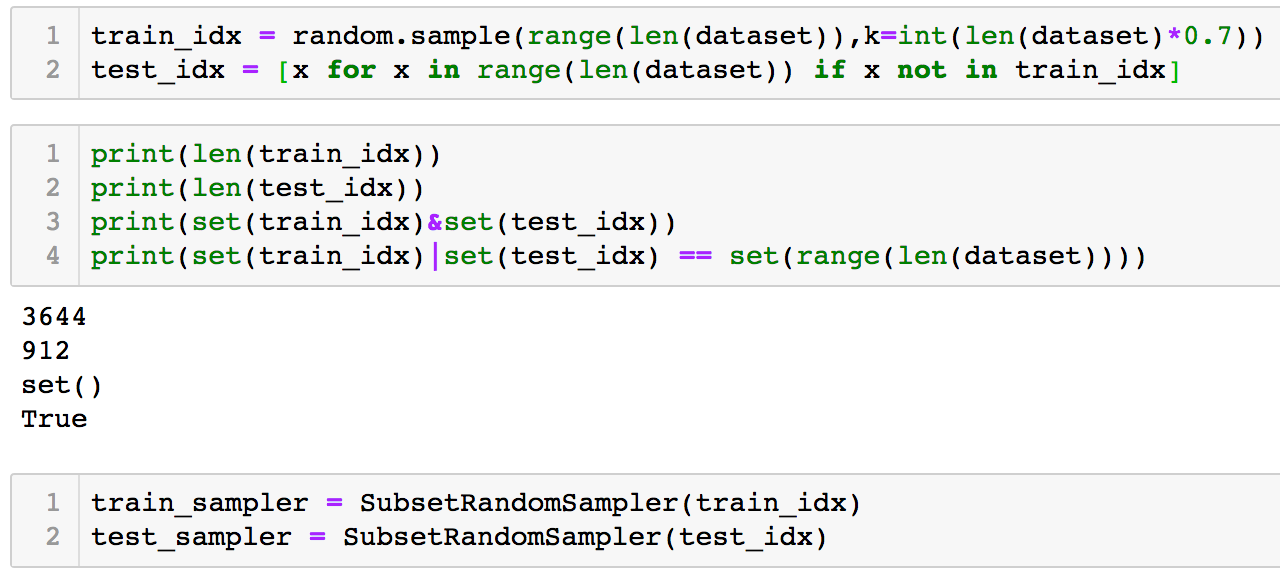
上面的代码中,我们先生成了 train_idx 和 test_idx,确保这两个集合没有交集,且来自全集的随机抽样;其次我们根据 train_idx 和 test_idx 得到了 train_sampler 和 test_sampler。
接着,我们构建 DataLoader 以供后续训练使用。
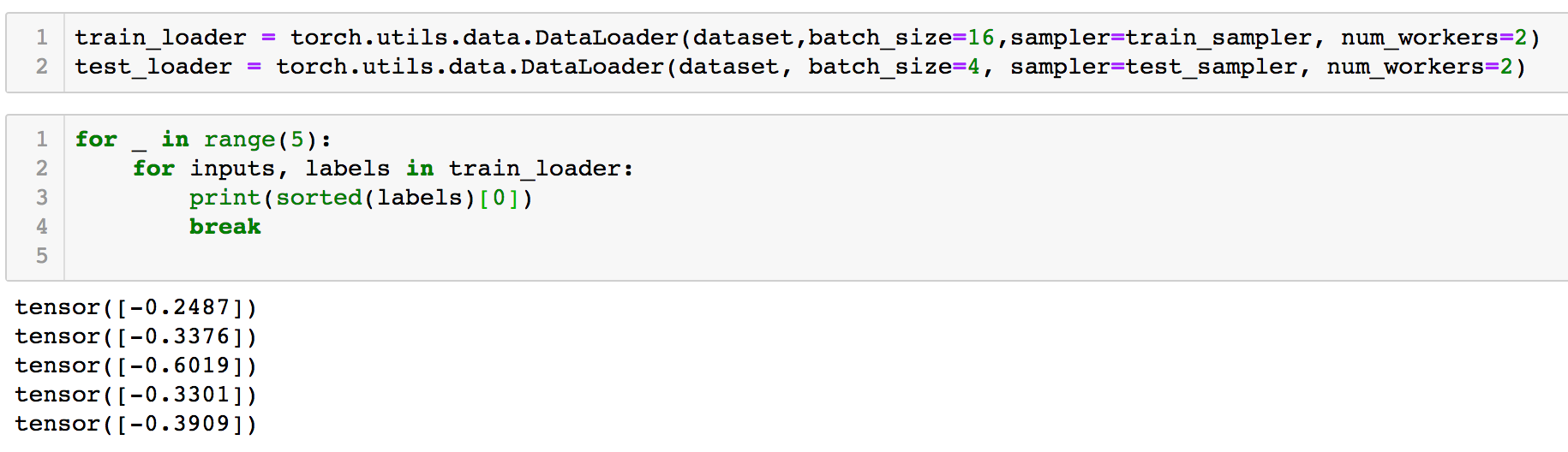
对于 train_loader 和 test_loader 我们分别设置其 batch_size 为 16 和 4,实际使用时需要对这一参数进行训练调整。可以看到,我们在枚举 DataLoader 中对数据时,每次的数据都不是一致的,这体现了 RandomSampler 的作用。
在数据输入处理完之后,我们需要构建网络架构了。为简化流程,我们采用以下代码中的架构:
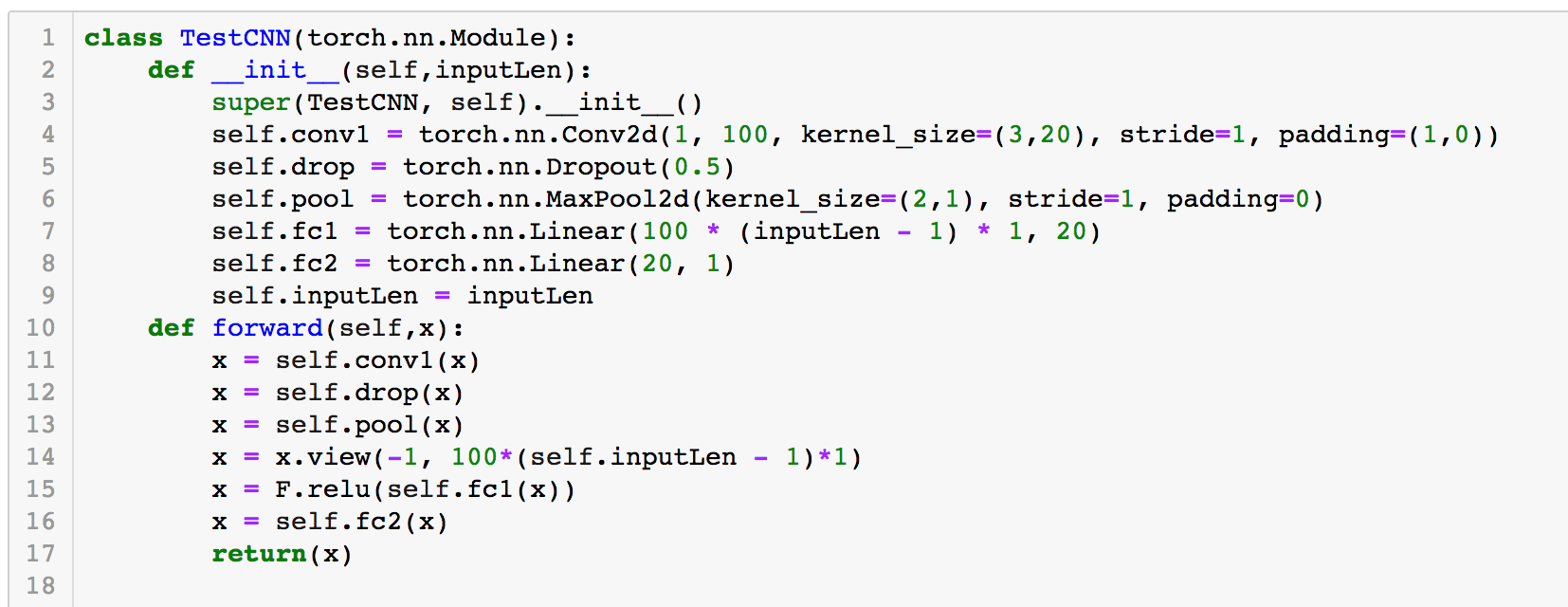
这一网络很简单,首先是一个 Conv2d 层,将输入的 features 做一个简单的卷积转换,其次经历一个 Dropout 层,防止训练过拟合;接着来一个 maxpool 层,以及两个线性的 full-connected 层,倒数第二层用了 RELU ,最后由于输出的不是概率值,所以直接采用线性输出。这一网络仅做教程演示,实际使用需根据情况进行修整。
数据搞定,网络搞定,接下来就要进行数据训练了。我们采用 Mean Square Error Loss 和 Adam optimizer。
下图为我们定义的 Train 函数。
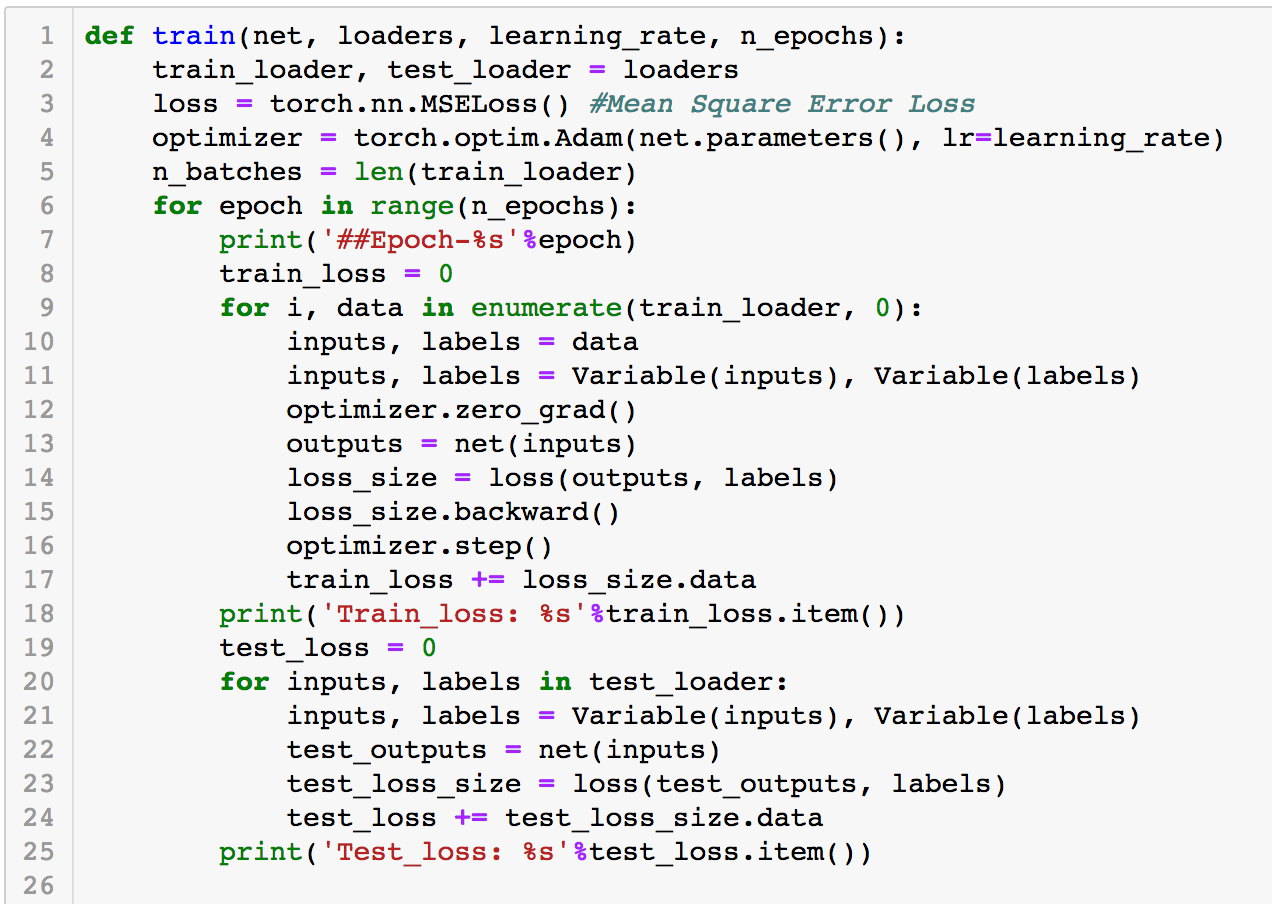
可以看到,我们在每个 epoch 中,计算了训练集的 loss 总和,同时使用当前的网络对 test 集合进行了评估。这样做的目的是可以直观的观察到模型是否在过拟合的路上越走越远。
实际在使用过程中,应该在训练过程中输出更多的信息,比如每隔 10% 就对当前模型的 Loss 进行输出,来观察模型训练过程中的进化过程。本文中为了减少冗余信息对此进行了简化和删减,读者在实际应用过程中应注意这一点。
另外,在本文中,对 Dev 集和 Test 集没有做过多的区分,实际在调参过程中,应该使用 Dev 集,再最终评估模型时应采用 Test 集,读者需注意这一点。
下图为文中代码的实际训练输出:
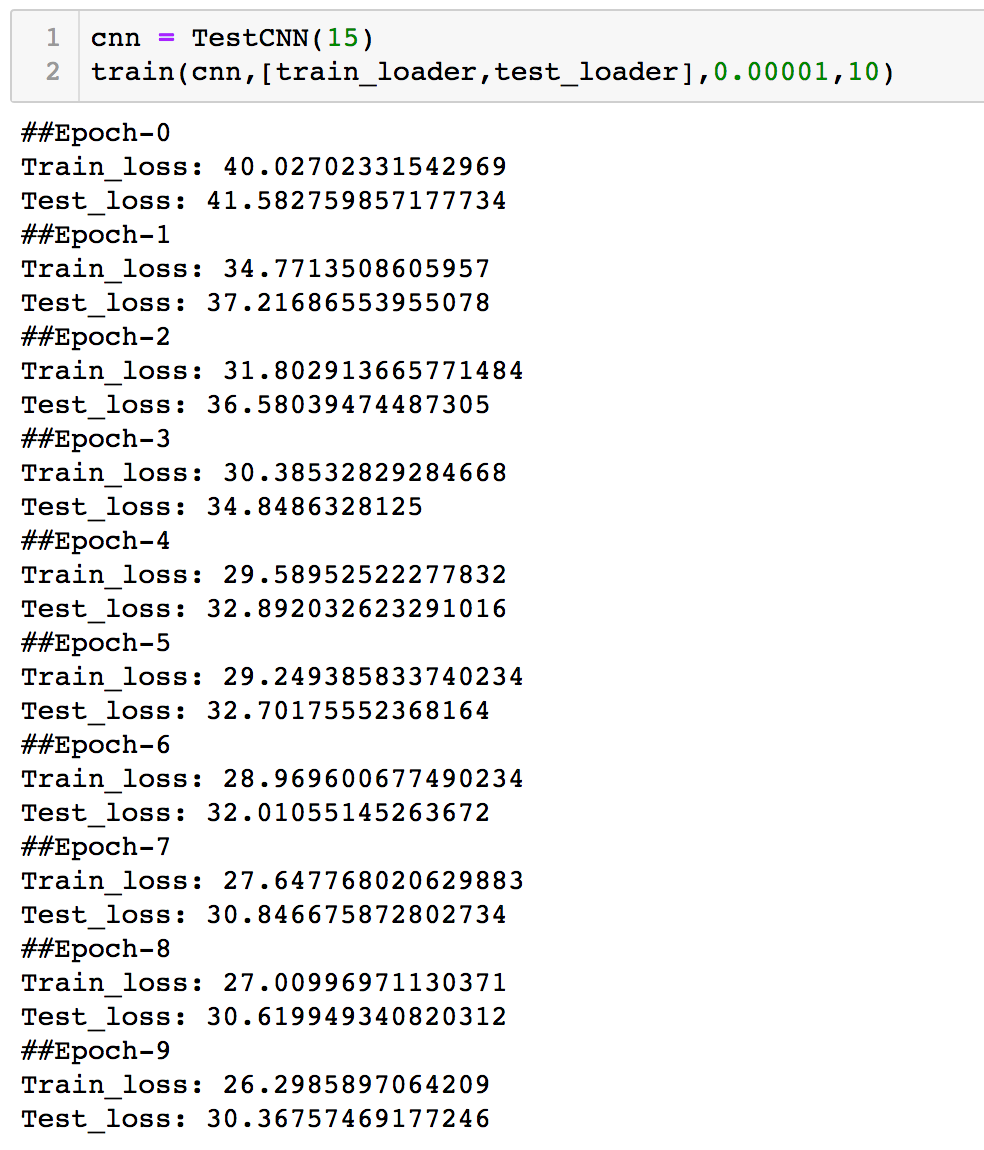
我们使用了 0.00001 的 learning_rate 和 10 的 epoch。可见训练过程中,Train 的 loss 在一直减少,这表明我们的梯度递减是 work 的;同时,Test 的 loss 也是递减,表明目前模型还未达到过拟合状态,但是很明显在最后几轮,Test 的 loss 下降幅度明显下降,值得关注。
最后,对于我们当前的场景,关注 Loss 并不直观,一个简单的方法是,查看预测值和已知值之间的相关性,若相关性较高,则表明模型较好,反之亦然。
下图中我们在 Test 集中,计算了预测值和实际值的相关系数,并绘图展示:
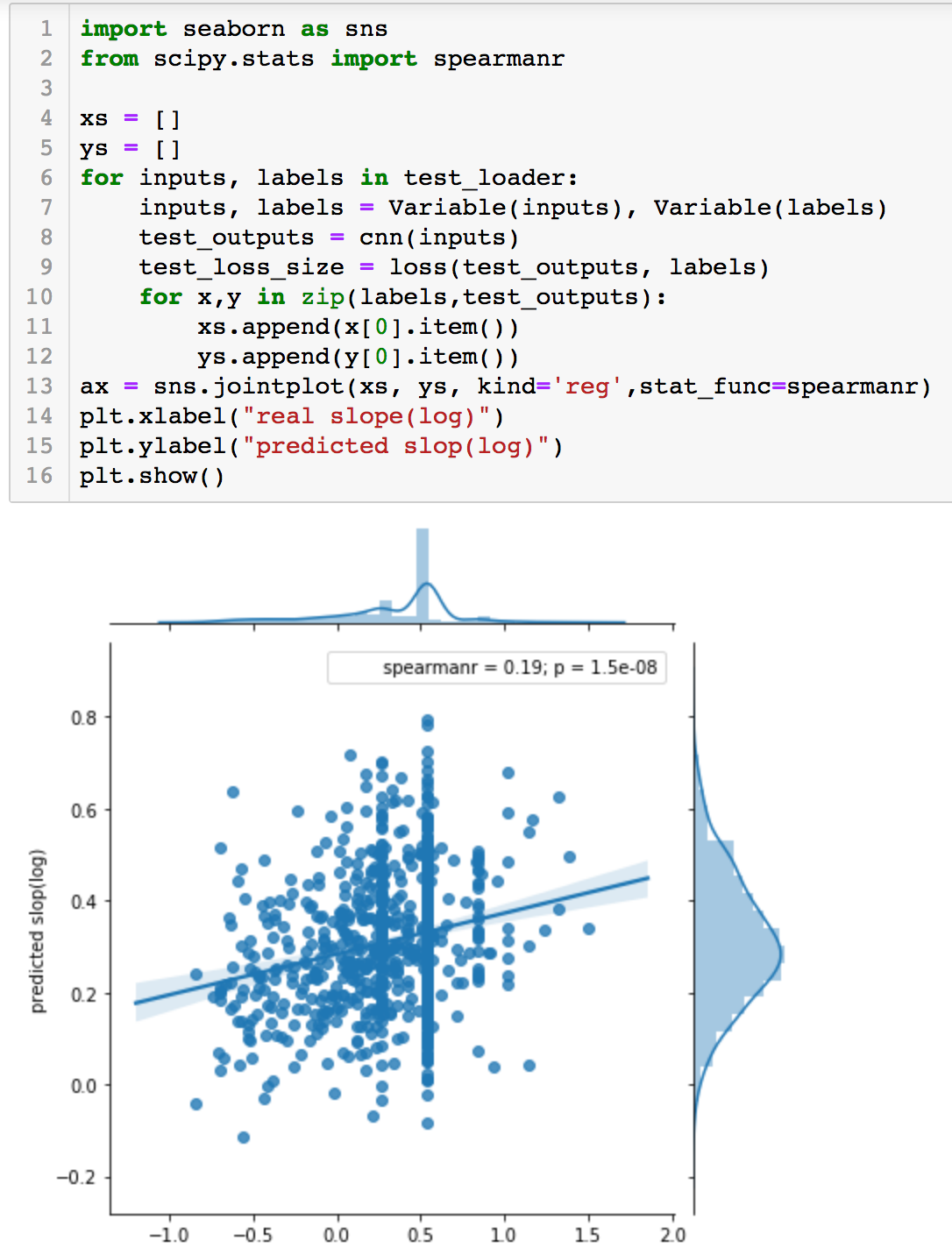
我们使用了 spearman 相关系数对结果进行了衡量,结果 0.19 并不是很好,说明网络、参数、数据输入等需要进一步的调整。大家可以用一些 Grid Search 的方法来进行参数调整,这里不再做进一步介绍。
转载请标注来源:
【老土译站】
2019-7-228
推荐阅读
您好,看到您这个简明教程很清晰,我想深入学习一下pytorch 在生物领域的应用,比如您这个蛋白质序列预测。 能否推荐一下容易入门的书籍和教程吗,针对生物方面的,感激不尽。
如果你是想入门生物信息学,可以搜一搜生物信息学的课程,coursera上有很多. 如果你要生物信息学中深度学习的应用,建议直接在pubmed网站进行搜索,这是一个例子https://www.ncbi.nlm.nih.gov/pubmed/?term=pytorch,直接看文献应该比看一些堆砌内容的书要强一些
你好,请问数据可以在哪里获取呢
可以搜索相关的文献,一般都会公开训练数据集
看完后受益匪浅。我能在自己的文章里引用您的代码吗?
当然不是全部。万分感谢。
文章是学术,而非商业使用。
谢谢您
当然可以 很荣幸
博主您好,特别感谢您提供的思路,请问这篇文章的源代码学习一下吗,具体想学习一下对于输入数据那里的处理,麻烦您了
可以试着搜一些类似工作的学术文章 (序列+神经网络),一般都附有公开的github代码可供参考